Su elevada tasa de reproducción es una de sus principales armas para sobrevivir a los antibióticos
El trabajo abre la puerta al desarrollo de medicamentos que puedan bloquear este mecanismo tan preciso
La elevada tasa de división de las bacterias, es decir, la exitosa forma en que se reproducen, es una de sus mejores armas contra los antibióticos. Un equipo del Consejo Superior de Investigaciones Científicas (CSIC) y la Universidad de Notre Dame (Estados Unidos) ha revelado la estructura de una maquinaria clave en ese proceso de división. Las conclusiones, publicadas en el último número de la revista Nature Communications, abren la puerta al diseño de un futuro medicamento capaz de bloquear esta maquinaria tan precisa, sin la cual las bacterias se vuelven sensibles al efecto antibiótico.
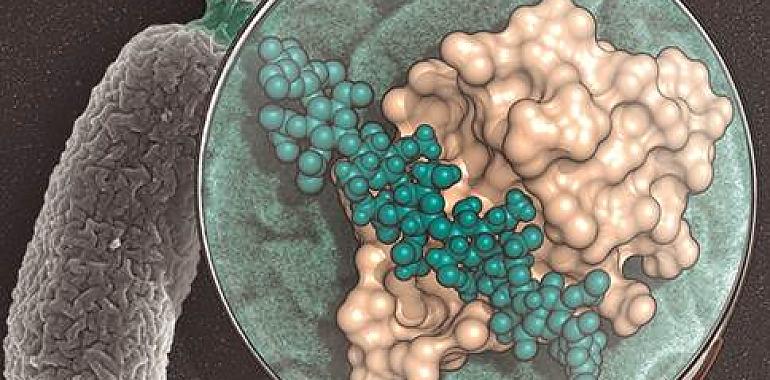
La división bacteriana está orquestada por un amplio conjunto de diversas proteínas que se ensamblan de manera ordenada y dinámica, formando una maquinaria precisa que garantiza el correcto devenir de la reproducción. Prácticamente todas las especies bacterianas poseen dominios especializados que reconocen la pared bacteriana (compuesta por peptidoglicano) en el momento de la división y posibilitan la correcta localización en el espacio y tiempo de estas proteínas durante la generación de las células hijas a partir de la célula madre. Los científicos han empleado la cristalografía de rayos X para obtener la estructura de esos dominios especializados.
En concreto, han estudiado el dominio SPOR (Sporulation-related repeat) de la proteína RlpA de Pseudomonas aeruginosa, una bacteria multiresistente para la cual existen muy pocos antibióticos disponibles, considerada por la Organización Mundial de la Salud como de “prioridad crítica”. “Los dominios SPOR son usados por casi todas las bacterias durante el proceso de división. Sin embargo, a pesar de su relevancia, nadie había podido hasta ahora clarificar cómo funcionaban durante la división. Todas estas proteínas reconocen el mismo tipo de pared que se produce durante la división bacteriana. Nuestro trabajo revela, por primera vez, por qué todos los dominios SPOR reconocen el mismo tipo de pared para facilitar la división”, explica el investigador del CSIC Juan Antonio Hermoso, del Instituto de Química Física Rocasolano.
La investigación propone un modelo extrapolable a todo tipo de bacterias. “Nuestro resultados aportan información a nivel atómico de cómo los dominios SPOR se unen a la pared bacteriana y, por tanto, abren la puerta al desarrollo de moléculas que puedan bloquear específicamente estos dominios, lo que volvería a las bacterias sensibles a los antibióticos”, destaca Hermoso.
Martín Alcorlo, David A. Dik, Stefania De Benedetti, Kiran V. Mahasenan, Mijoon Lee, Teresa DomínguezGil, Dusan Hesek, Elena Lastochkin, Daniel López, Bill Boggess, Shahriar Mobashery y Juan A. Hermoso. Structural basis of denuded glycan recognition by SPOR domains in bacterial cell division. Nature Communications. DOI: 10.1038/s41467-019-13354-4
Foto: Dominio SPOR (en color beige) unido a la pared bacteriana (en esferas verdes). En el fondo se representa una imagen de microscopía de Pseudomonas aeruginosa dividiéndose en dos./ IFQR-CSIC